SARS-CoV-2, nuovo approccio consente di diagnosticare se il virus è ‘vivo’ e si sta replicando

Uno studio condotto dal Cnr-Ibiom di Bari insieme all’Università di Bari e all’Università Statale di Milano, con il supporto della piattaforma bioinformatica e genomica di Elixir Italia, ha sviluppato una metodologia che consente di verificare il grado di infettività di una persona affetta da Covid-19, discriminando tra il genoma a RNA del virus e le molecole derivanti dalla sua trascrizione. Il lavoro è pubblicato su Communications Biology

Roma, 5 ottobre 2021 – Un team dell’Istituto di biomembrane, bioenergetica e biotecnologie molecolari del Consiglio nazionale delle ricerche (Cnr-Ibiom) di Bari, dell’Università degli Studi di Bari “Aldo Moro”, dell’Università Statale di Milano, dell’Istituto Zooprofilattico Sperimentale di Puglia e Basilicata e del Laboratorio Covid dell’Ospedale “Di Venere” di Bari, con il supporto della piattaforma genomica e bioinformatica messa a disposizione dal nodo italiano dell’Infrastruttura di ricerca europea Elixir per le scienze della vita, ha effettuato uno studio su 166 soggetti affetti da Covid-19 con differente grado di carica virale nel quale è stata messa a punto una metodologia per determinare il numero assoluto di molecole di RNA virale contenute nei tamponi molecolari utilizzati per individuare la positività al virus. La ricerca pubblicata su Communications Biology permette di individuare il grado di infettività di persona affetta da Covid-19.
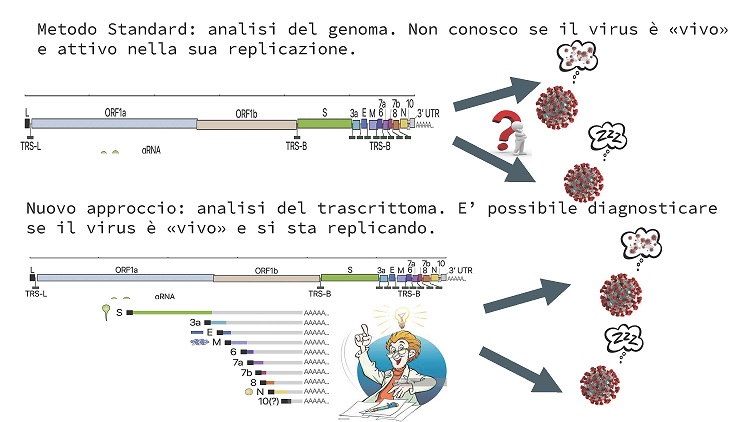
A seguito dell’infezione da SARS-Cov-2, il virus produce due tipi di molecole di RNA: 1) un filamento di RNA di circa 30,000 nucleotidi corrispondente al genoma completo del virus; 2) una serie di molecole di RNA discontinue dette anche trascritti-sub-genomici che codificano per le proteine necessarie ad assemblare nuovi virioni e sono necessari per la replicazione del virus. Queste molecole costituiscono dunque un indice dell’attività di replicazione virale e, indirettamente, del grado di infettività di un soggetto affetto da Covid-19.
“La nuova metodologia sviluppata, basata sull’utilizzo della tecnica della “droplet digital PCR” (ddPCR) consente di conteggiare separatamente il numero di molecole di RNA genomiche e subgenomiche. I test molecolari standard attualmente utilizzati, basati invece sulla tecnica della “real time PCR” non sono in grado di discriminare tra i due tipi di RNA virali”, spiega Graziano Pesole del Cnr-Ibiom.
Dal momento che le molecole subgenomiche sono marcatori di un processo infettivo in corso, nel quale si ha proliferazione di nuove particelle virali, approcci basati su questo principio potranno essere applicati in futuro per determinare il grado di infettività di una persona, anche nel corso del tempo.
“Lo studio ha mostrato che la percentuale di RNA subgenomici è correlata alla carica virale ed è anche analogamente determinabile da analisi mediante sequenziamento massivo del trascrittoma. I risultati presentati contribuiscono a comprendere meglio la dinamica dell’espressione di SARS-Cov-2 in diverse condizioni e a mettere a punto strategie diagnostiche innovative per fronteggiare la pandemia da SARS-Cov-2”, conclude Pesole.